A través de mapas es posible visualizar el comportamiento de una variable en un determinado espacio. Con Leaflet, en R, pueden obtenerse mapas con facilidad y rapidez.
Para instalar este paquete, simplemente se hace uso del comando install.packages. Luego se activa con la orden library.
Al escribir los siguientes comandos, se obtiene una representación global de los 5 continentes.
Las funciones setView(), fitBounds, flyTo() sirven para enfocar áreas de interés específico. Por ejemplo:
Es preciso introducir latitud, longitud y zoom, este último para controlar el acercamiento. Es posible hacer mapas de áreas más específicas, si se cuenta con mediciones. Los mapas que aparecen seguidamente, a modo de ilustración, fueron obtenidos a partir de data ficticia de 3 direcciones de la ciudad, escogidas arbitrariamente, y haciendo uso de la aplicacion Nominatim de Open Street Map.
La aplicación admite una solicitud por segundo, según la documentación, y su dirección web es:
A esa dirección web deben añadirse la dirección del lugar, cuyas coordenadas estamos requiriendo, y posteriormente una sección que corresponde al formato de la data. Uno modo de descargar varias coordenadas a la vez es el siguiente, aunque existen paquetes que podrían facilitar aún más este procedimiento.
Ahora con lapply se puede hacer un bucle que ponga esas 3 direcciones en el formato requerido.
Finalmente para descargar las coordenadas;
Con el operador `$` pueden extraerse subconjuntos de la data, p.e:
El $`bbox` representa un bounding box, es decir un rectangulo en cuya área se encuentra el sitio de interés. Usaré la data contenida en la lista ox para obtener algunos mapas.
Entonces al introdocir las coordenadas en la función flyTo() se obtiene un mapa de la localidad:
En el caso anterior, pudo usarse setView(), en ese caso esas funciones serían intercambiables.
Para añadir marcadores indicando la ubicación del lugar se usa addMarkers, si se desea cambiar el icono, por otro que se considere más apropiado para el lugar que se pretende señalar, se hace uso del argumento icon.
La función addRectangles sirve para señalar áreas que puedan ser de interés.
En ocasiones es necesario representar en el espacio la frecuencia o intensidad de un fenómeno o variable. Un ejemplo con data simulada:
Con addProviderTiles se modifica el formato de los mapas. Existen diversos formatos, sin embargo, algunos requieren registro.
Otro modo de representar intensidad es a través de los llamados heatmaps. Uno puede obtenerlos facilmente cargando leaflet.extras.
Otro modo de producir estos heatmaps involucra generar líneas de contorno o circulos concentricos que den una idea de intensidad gradual de un fenómeno a medida que se avanza hacía el centro de un área, por ejemplo el siguiente código
Teniendo lo anterior, faltaría seleccionar colores mediante una palette, el paquete ofrece 3 funciones principales para crear palettes: colorBin, colorFactor y colorNumeric:
Esto permite dibujar el mapa:
Un ejemplo más con otra región. Supongamos que se cuenta con una data referente a presión atmosférica de una regíon del Caribe, por ejemplo las llamadas Antillas Menores, cuyas coordenadas son 14°14' 61°21'.
La expectativa es que la coloración dé una idea de la mayor o menor intensidad de la presión. El aspecto nocturno lo da el "Esri.WorldImagery" en la función addProviderTiles().
Para mapas temáticos se precisa contar con alguna data georreferenciada. Algunos sitios permiten descargar datos de este tipo.
La función colorFactor permite la creación de una paleta de colores para identificar variables de tipo factor.
Lo siguiente es solo para que el texto de la variable ECONOMY aparezca en español.
La documentación recomienda que las etiquetas estén en HTML es la razón del uso de htmltools
El resultado es un mapa interactivo, en el sentido de que si alguién mueve el cursor encima de un área especifica debe surgir información asociada a dicha área en la etiqueta (label), aquí no se puede apreciar el efecto porque no encontré un modo de subir el archivo de otra manera que no fuera una imagen fija.
En fin, el paquete ofrece formas rápidas de obtener mapas, y además también los recursos para añadir la información que se quiera representar con los efectos estéticos que se considerén adecuados, según sea el caso.
library(leaflet) library(leaflet.extra) # heatmaps library(magrittr) # para haceer uso del pipeline '%>%' leaflet() %>% addTiles()<
Las funciones setView(), fitBounds, flyTo() sirven para enfocar áreas de interés específico. Por ejemplo:
leaflet() %>% addTiles() %>%
setView(lng = -66.9, lat = 10.5,
zoom = 11)Es preciso introducir latitud, longitud y zoom, este último para controlar el acercamiento. Es posible hacer mapas de áreas más específicas, si se cuenta con mediciones. Los mapas que aparecen seguidamente, a modo de ilustración, fueron obtenidos a partir de data ficticia de 3 direcciones de la ciudad, escogidas arbitrariamente, y haciendo uso de la aplicacion Nominatim de Open Street Map.
La aplicación admite una solicitud por segundo, según la documentación, y su dirección web es:
opsm_url <- "https://nominatim.openstreetmap.org/search/"
A esa dirección web deben añadirse la dirección del lugar, cuyas coordenadas estamos requiriendo, y posteriormente una sección que corresponde al formato de la data. Uno modo de descargar varias coordenadas a la vez es el siguiente, aunque existen paquetes que podrían facilitar aún más este procedimiento.
especs <- "?format=json&addressdetails=0&limit=1"
direcs <- c("Av Francisco Solano, Sabana Grande, El Recreo, Libertador, Caracas",
"Av. Eugenio Mendoza, La Castellana, Chacao, Caracas",
"Estación Bello Monte, Municipio Baruta, Caracas, Venezuela")Ahora con lapply se puede hacer un bucle que ponga esas 3 direcciones en el formato requerido.
comUrls <- lapply(direcs, function(x) paste0(opsm_url,
gsub("\\s+",
"\\%20",
x),
especs))Finalmente para descargar las coordenadas;
geolocs <- lapply(comUrls,
function(x) {d <- jsonlite::fromJSON(x)
Sys.sleep(3)
return(list(data.frame(lon = as.numeric(d$lon),
lat = as.numeric(d$lat)),
bbox = d$boundingbox))})
ox <- set_names(geolocs, c("Fco. Solano",
"La Castellana",
"Est. Bello Monte"))Con el operador `$` pueden extraerse subconjuntos de la data, p.e:
ox$`Est. Bello Monte` # $`Est. Bello Monte` # $`Est. Bello Monte`[[1]] # lon lat # 1 -66.87465 10.48731 # $`Est. Bello Monte`$bbox # $`Est. Bello Monte`$bbox[[1]] # [1] "10.482309" "10.492309" "-66.8796527" "-66.8696527"
El $`bbox` representa un bounding box, es decir un rectangulo en cuya área se encuentra el sitio de interés. Usaré la data contenida en la lista ox para obtener algunos mapas.
Entonces al introdocir las coordenadas en la función flyTo() se obtiene un mapa de la localidad:
leaflet() %>%
addTiles() %>%
flyTo(lng = -66.87465, lat = 10.48731, zoom = 17) %>%
addMarkers(lng = -66.87465, lat = 10.48731,
label = "Est. Bello Monte")En el caso anterior, pudo usarse setView(), en ese caso esas funciones serían intercambiables.
Para añadir marcadores indicando la ubicación del lugar se usa addMarkers, si se desea cambiar el icono, por otro que se considere más apropiado para el lugar que se pretende señalar, se hace uso del argumento icon.
leaflet() %>%
addTiles() %>%
flyTo(lng = -66.87465, lat = 10.48731, zoom = 17) %>%
addMarkers(lng = -66.87465, lat = 10.48731,
label = "Est. Bello Monte") La función addRectangles sirve para señalar áreas que puedan ser de interés.
mlon <- mean(c(-66.8511152, -66.8809526))
mlat <- mean(c(10.4949489, 10.4966085))
leaflet() %>%
addTiles() %>%
setView(lng = mlon, lat = mlat,
zoom = 14) %>%
addRectangles(lng1 = -66.8511152,
lat1 = 10.4949489,
lng2 = -66.8809526,
lat2 = 10.4966085) %>%
addRectangles(lng1 = -66.8796527,
lat1 = 10.482309,
lng2 = -66.8696527,
lat2 = 10.492309)En ocasiones es necesario representar en el espacio la frecuencia o intensidad de un fenómeno o variable. Un ejemplo con data simulada:
n1 <- 20
set.seed(1)
df2 <- data.frame(lon = rnorm(n1, mean = -66.88095, sd = 10^-3),
lat = rnorm(n1, mean = 10.49661, sd = 10^-3),
dims = runif(n1, min = 2, max = 10))
leaflet(df2) %>%
addTiles() %>%
setView(lng = -66.88095, lat = 10.49661,
zoom = 17) %>%
addCircleMarkers(lng = ~lon, lat = ~lat,
radius = ~sqrt(dims) * 7,
fillColor = "#FF4500",
opacity = .9,
color = "transparent",
fillOpacity = .7) %>%
addProviderTiles(providers$Stamen.Toner)Con addProviderTiles se modifica el formato de los mapas. Existen diversos formatos, sin embargo, algunos requieren registro.
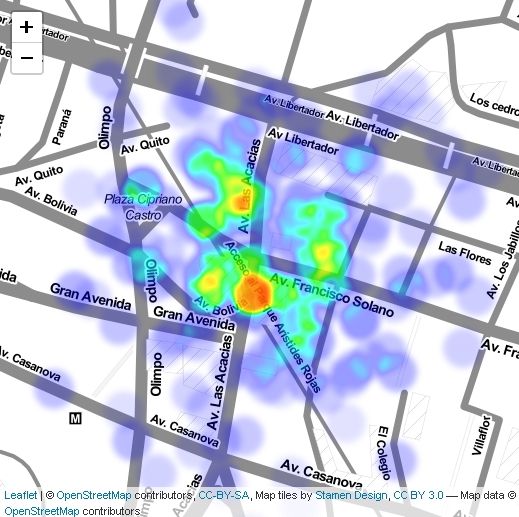
Heatmaps
Otro modo de representar intensidad es a través de los llamados heatmaps. Uno puede obtenerlos facilmente cargando leaflet.extras.
library(leaflet.extras)
n1 <- 200
set.seed(1)
df2 <- data.frame(lon = rnorm(n1, mean = -66.88095, sd = 10^-3),
lat = rnorm(n1, mean = 10.49661, sd = 10^-3),
dims = rpois(n1, 20))
leaflet(df2) %>%
addTiles() %>%
setView(lng = -66.88095, lat = 10.49661,
zoom = 17) %>%
addHeatmap(lng = ~lon, lat = ~lat,
intensity = ~dims,
max = 100,
radius = 20,
blur = 10) %>%
addProviderTiles(providers$Stamen.TonerLite)Otro modo de producir estos heatmaps involucra generar líneas de contorno o circulos concentricos que den una idea de intensidad gradual de un fenómeno a medida que se avanza hacía el centro de un área, por ejemplo el siguiente código
library(KernSmooth)
library(sp)
kde <- KernSmooth::bkde2D(df2[, c("lon", "lat")],
bandwidth=c(.0045, .0068),
gridsize = c(100,100))
CL <- contourLines(kde$x1 , kde$x2 , kde$fhat)
# extraer niveles de las líneas.
LEVS <- as.factor(sapply(CL, `[[`, "level"))
NLEV <- length(levels(LEVS))
## convertir líneas de contorno a poligonos.
pgons <- lapply(1:length(CL), function(i)
sp::Polygons(list(sp::Polygon(cbind(CL[[i]]$x,
CL[[i]]$y))),
ID=i))
spgons <- sp::SpatialPolygons(pgons)
dat <- data.frame(ID = as.character(1:13),
Levs = LEVS)
df <- sp::SpatialPolygonsDataFrame(spgons, dat)
df@data$levsn <- sapply(CL,`[[`, "level")Teniendo lo anterior, faltaría seleccionar colores mediante una palette, el paquete ofrece 3 funciones principales para crear palettes: colorBin, colorFactor y colorNumeric:
p2 <- colorBin("YlOrRd", 5, domain = sapply(CL,`[[`, "level"),
na.color = "transparent")>Esto permite dibujar el mapa:
df %>%
leaflet() %>%
addTiles() %>%
addPolygons(color = ~p2(df@data$levsn)) %>%
addProviderTiles(providers$Stamen.Terrain) %>%
addLegend(position = "topright",
pal = p2,
values = df@data$levsn)Un ejemplo más con otra región. Supongamos que se cuenta con una data referente a presión atmosférica de una regíon del Caribe, por ejemplo las llamadas Antillas Menores, cuyas coordenadas son 14°14' 61°21'.
latAnt <- 14 + 14/60
lonAnt <- -1 * (61 + 21/60) n <- 100
set.seed(1)
dfx <- data.frame(lon = rnorm(n,
mean = lonAnt,
sd = 1.3),
lat = rnorm(n,
latAnt,
sd = 1.5),
presion = rpois(n, 1000))
leaflet(dfx) %>%
addTiles() %>%
setView(lng = lonAnt,
lat = latAnt,
zoom = 5) %>%
addHeatmap(lng = ~lon, lat = ~lat,
intensity = ~presion,
max = 100,
radius = 20,
blur = 30) %>%
addProviderTiles("Esri.WorldImagery")La expectativa es que la coloración dé una idea de la mayor o menor intensidad de la presión. El aspecto nocturno lo da el "Esri.WorldImagery" en la función addProviderTiles().
Choropleths
Para mapas temáticos se precisa contar con alguna data georreferenciada. Algunos sitios permiten descargar datos de este tipo.
URL <- "https://www.naturalearthdata.com/downloads/110m-cultural-vectors/110_cultural.zip"
library(sf)
q <- "SELECT NAME_ES, ECONOMY
FROM \"ne_110m_admin_0_map_units\"
WHERE REGION_UN IN ('Europe', 'Africa')
AND NAME_ES NOT IN ('Rusia', 'Svalbard')
OR SUBREGION = 'Western Asia'
AND GDP_YEAR = 2016"
m2 <- st_read(f1, query = q)La función colorFactor permite la creación de una paleta de colores para identificar variables de tipo factor.
Lo siguiente es solo para que el texto de la variable ECONOMY aparezca en español.
m2$economia <- ifelse(substr(m2$ECONOMY, 1, 1) == '1',
"1. Reg.Desarrollada G7",
ifelse(substr(m2$ECONOMY, 1, 1) == '2',
"2. Reg.Desarrollada No G7",
ifelse(substr(m2$ECONOMY, 1, 1) == '4',
"4. Reg.Emergente MIKT",
ifelse(substr(m2$ECONOMY, 1, 1) == '5',
"5. Reg.Emergente G20",
ifelse(substr(m2$ECONOMY, 1, 1) == '6',
"6. Reg.En Desarrollo",
"7. Reg.Menos Desarrollada")))))La documentación recomienda que las etiquetas estén en HTML es la razón del uso de htmltools
library(htmltools)
meanlon <- mean(c(st_bbox(m2)[1], st_bbox(m2)[3]))
meanlat <- mean(c(st_bbox(m2)[2], st_bbox(m2)[4]))
m2 %>%
leaflet() %>%
addTiles() %>%
setView(lng = meanlon, lat = meanlat,
zoom = 3) %>%
addPolygons(stroke = FALSE,
fillColor = ~factpal(economia),
label = ~htmlEscape(NAME_ES),
color = "white",
fillOpacity = 0.5) %>%
addLegend(position = "bottomleft", factpal,
values = ~economia,
opacity = .9,
title = "Tipo de Economía")
El resultado es un mapa interactivo, en el sentido de que si alguién mueve el cursor encima de un área especifica debe surgir información asociada a dicha área en la etiqueta (label), aquí no se puede apreciar el efecto porque no encontré un modo de subir el archivo de otra manera que no fuera una imagen fija.
En fin, el paquete ofrece formas rápidas de obtener mapas, y además también los recursos para añadir la información que se quiera representar con los efectos estéticos que se considerén adecuados, según sea el caso.









Comentarios
Publicar un comentario